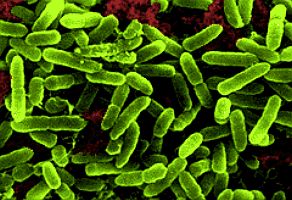
Investigadores de la Universidad de Liverpool están estudiando la evolución de Pseudomonas aeruginosa en el tiempo en los pulmones de diez personas con fibrosis quística para entender por qué persiste en estos pacientes y por qué su respuesta a los antibióticos es tan variada.
 No hay que alejarse de las bacterias: que respiramos miles de ellos cada día. La mayoría de ellos no nos hacen mucho daño, pero algunas – como ciertas cepas de Streptococcus, Staphylococcus y Pseudomonas – tienen el potencial de causar infecciones graves como la neumonía y pleuresía, si se meten en nuestros pulmones.
No hay que alejarse de las bacterias: que respiramos miles de ellos cada día. La mayoría de ellos no nos hacen mucho daño, pero algunas – como ciertas cepas de Streptococcus, Staphylococcus y Pseudomonas – tienen el potencial de causar infecciones graves como la neumonía y pleuresía, si se meten en nuestros pulmones.
Afortunadamente, la mayoría de las personas sanas tienen una serie de barreras mecánicas y biológicas – como el moco y los cilios en el tracto respiratorio (nuestra escalera mecánica mucociliar) – que evitan que las bacterias que inhalamos lleguen a nuestros pulmones.
Para las personas con fibrosis quística, sin embargo, es una historia diferente. Una mutación genética significa que producen una secreción espesa y pegajosa que recubre el tracto respiratorio, afectando gravemente su escalera mecánica mucociliar. En consecuencia, las bacterias dañinas pueden entrar en los pulmones y permanecer allí, con seguridad dentro de capullos de protección o bolsas, lo que hacen los polisacáridos secretores.
«Una vez que estas bacterias colonizan los pulmones de una persona con fibrosis quística, que es imposible deshacerse de ellos por completo con antibióticos», dice el profesor Craig Winstanley en el Instituto de la infección y la Salud Global de la Universidad de Liverpool. Las bacterias permanecen dentro de los pulmones de la persona para el resto de su vida, haciendo que las infecciones pulmonares graves maten a las personas con fibrosis quística.
Hasta hace unos 15 años atrás, los médicos creían que estas cepas no eram transmisible entre las personas con fibrosis quística. En 1996, sin embargo, Craig y sus colegas identificaron una cepa de Pseudomonas aeruginosa, que no sólo causa infecciones particularmente graves en las personas que lo capturan, pero también podría transmitirse de paciente a paciente.
«Los niños con fibrosis quística utilizan para ir a los campamentos de verano juntos, pero una vez la gente se dio cuenta de que podía pasar cepas desagradables de las infecciones pulmonares en el uno al otro, por supuesto que tenía que parar», dice. «En nuestra clínica aquí, separar los pacientes con fibrosis quística – vienen en el tratamiento en días distintos – por lo que no puede pasar en las cepas virulentas entre sí.»
Debido a que esta cepa particular de P. aeruginosa se identificó por primera vez en Liverpool, Craig y sus colegas nombraron el ‘Liverpool cepa epidémica «(LES). Se desarrolló una prueba rápida de diagnóstico para identificar LES en la saliva tomadas de las personas con fibrosis quística, con base en la secuencia de ADN en determinadas regiones de su genoma (un procedimiento conocido como un ensayo de tipificación molecular).
Sin embargo, a pesar de que ahora se puede identificar rápidamente la presión del EEI, los médicos han quedado desconcertados por el hecho de que los antibióticos destinados a la cepa tener efectos radicalmente diferentes en los distintos pacientes – incluso entre las personas con la misma infección. «Es al azar si un antibiótico en particular va a funcionar», dice Craig. «Me temo que aún estamos en la Edad Media a la hora de tratar las infecciones en estos pacientes.
«A veces un paciente se pone mejor, a veces no, y no sabemos por qué. Ni siquiera sé lo que los antibióticos están haciendo a las infecciones, biológicamente. Ciertamente no son la eliminación de ellos.»
¿Qué pasa si
Para tratar de entender este dilema, Craig se hizo la pregunta más importante que tan a menudo lleva a los avances científicos: ¿qué pasaría si? ¿Qué pasa si los antibióticos tienen efectos tan variados en las infecciones LES debido a las fuerzas evolutivas que actúan sobre las bacterias en los pulmones?
«La idea predominante es que las infecciones en los pacientes con FQ son causados por una sola bacteria, genéticamente uniformes», dice. «Los microbiólogos tomar una muestra de esputo del paciente y suelen aislar una bacteria P. aeruginosa en la muestra. A continuación, comprobar que solo aislamiento para los que los antibióticos podrían ser efectivos contra ella y enviar la respuesta a los médicos.»
Este planteamiento es perfectamente válido para una infección como una quemadura, que cura (y mata a las bacterias que infectan) rápidamente en las personas sanas, pero las bacterias que entran en los pulmones de las personas con fibrosis quística viven allí desde hace años. ¿Qué sucede si, durante ese tiempo, la bacteria infectante original comenzó a mutar, la producción de clones que fueron genéticamente diferente de la «madre»? ¿Podría ser genéticamente diferentes versiones de la vida cepa LES en personas con fibrosis quística – contables para los diversos efectos de los antibióticos dirigidos contra la bacteria de la original «padre»?
Para probar esta hipótesis, Craig decidió a mirar a varias personas con fibrosis quística con la misma infección durante un período de tiempo. En 2009, él y sus colegas tomaron muestras de esputo a intervalos mensuales de cada diez personas con fibrosis quística infectados con la misma cepa de P. aeruginosa LES.
Los investigadores aislaron y crecieron clones de no uno, sino 40 las bacterias enteros – todos los representantes de LES – de todas las muestras de esputo, lo que resulta en un total de 1720 cepas o clones. A continuación, se caracteriza cada uno de estos clones de 1720 por 15 rasgos o comportamientos medibles (fenotipos).
Estos incluyen lo que cada clon realidad parecía en una placa de Petri (su color y morfología), si puede crecer en ciertos tipos de medios de comunicación, lo que (en su caso) los antibióticos es resistente a, lo que las toxinas que segrega y si había perdido la capacidad de utilizar la glucosa como su única fuente de carbono. También hicimos algunas pruebas genéticas, ayudado por el Dr. Steve Paterson en el Centro de la Universidad para la Investigación Genómica, para buscar fragmentos específicos de ADN y fragmentos de los cromosomas que eran inestables.
El uso de estas 15 características o fenotipos, se elaboró un perfil de cada uno de estos clones de 1720 y los compararon para ver si eran idénticos o podría ser clasificado como diferentes tipos de la misma cepa.
Los resultados, publicados en el Diario Americano de Medicina Respiratoria y Cuidados Críticos en 2011, reveló una cantidad sorprendente de la diversidad a través de estos 15 rasgos. «Hemos encontrado cientos de subtipos distintos de la misma cepa. Mucho más que probable que la gente había imaginado», dice Craig. Lo más sorprendente de todo, la mayor parte de esa diversidad fenotípica o de comportamiento se produjo en los pacientes individuales en lugar de ser debido a las diferencias entre los pacientes.
Las bacterias se habían sometido a rápidas mutaciones genéticas (en un período de meses) en el pulmón que les permitió cambiar su comportamiento. Esto explica la variada respuesta a los antibióticos y otros tratamientos visto hasta la fecha, y por qué es tan difícil decidir cuál es el antibiótico a utilizar para un resultado exitoso.
«De hecho», dice Craig, «las pruebas actuales no tiene ningún sentido, a pesar de que está ampliamente utilizado. Y todo el mundo está empezando a reconocer esto, pero nadie sabe cuáles son las alternativas. Lo que no ayuda es no reconocer que esta que pasa «.
«Teniendo en 40 colonias individuales pequeñas por muestra, y teniendo un montón de muestras en el tiempo es lo que realmente puso de manifiesto que la diversidad sorprendente. Se ha dado una perspectiva completamente nueva sobre estas infecciones», dice Steve.
Debido a que estos cambios en las bacterias son claramente muy importantes, lo que les permite sobrevivir y prosperar en personas con fibrosis quística, que es crucial para comprender cómo, cuándo y por qué ocurren.
Para obtener un mejor manejo de las mutaciones – y los cambios biológicos que generan – Steve y Craig ahora tienen un período de tres años de Wellcome Trust, proyecto de subvención para acercar más precisamente en los aislamientos obtenidos en 2009. Se estará colaborando con el Dr. Mike Brockhurst en el Instituto de Biología Integrativa, que será la aplicación de técnicas de genética de poblaciones y la biología evolutiva para el conjunto de datos.
Steve, Mike y sus colegas van a utilizar la próxima generación de alto rendimiento de secuenciación de ADN de tecnología para secuenciar el cromosoma completo de los lotes representativos de los aislados a partir de diez pacientes de la muestra de Craig en 2009.
El Centro de Investigación Genómica en el Liverpool cuenta con una SOLiD Ciencias de la Vida 5500 de la máquina XL – el primero de su tipo en el Reino Unido. «Funciona 250 000 veces más rápido que la tecnología utilizada para secuenciar el genoma humano hace diez años», dice Steve. «Se puede leer 30 mil millones de letras de la secuencia de ADN al día, frente a cuatro mil millones utilizando las máquinas actuales. Eso es el equivalente de alrededor de 100 genomas humanos en cinco días. Así que voy a ser capaz de secuencia de alrededor de 100 genomas bacterianos en una semana.
La tecnología les permitirá investigar las mutaciones en las bacterias LES con una resolución mucho más fina de lo que nadie ha sido capaz de hacer antes. «Podemos bacterias secuencias múltiples y comparar las cosas a los cambios individuales en los pares de bases en las secuencias de genes.»
La predicción de la evolución
Un aspecto importante y singular del proyecto es que es la primera vez que ha sido la evolución de la LES seguido en personas reales, vivas, en lugar de en el laboratorio, en tubos de ensayo y de los sistemas artificiales. Lo que más excita a Steve es el hecho de que debido a que cada una de las diez personas de la cual las muestras fueron recolectadas tenía la misma cepa de LES, su evolución ha sido creado de forma paralela, pero independiente.
Dado que las bacterias se reproducen rápidamente (cada media hora o algo así), Steve será capaz de ver las mutaciones que ocurren en el espacio de meses o semanas. A raíz de las mutaciones que se producen deben ayudar a arrojar luz sobre cómo la bacteria se adapta de manera tan eficiente a las personas con fibrosis quística y los cambios en la cuenta de las poblaciones de bacterias para su éxito.
«Estos pacientes están siendo afectados por una gran cantidad de antibióticos como parte de su terapia estándar, por lo que los errores de desarrollar una respuesta a eso. Vamos a tratar de encontrar qué mutaciones permiten que estos errores para convertirse en resistentes a los antibióticos o los cambios que les permiten segregar toxinas en los pulmones «, dice Steve.
También tratará de establecer los cambios o fluctuaciones en la composición de la población bacteriana que puede hacer que la infección más virulenta o desencadenar una exacerbación en las personas. Una gran cantidad de daños se hace a los pulmones durante estas exacerbaciones, y los pacientes tienen que ser hospitalizados y recibir antibióticos por vía intravenosa.
La duración de la infección – y su evolución en diez ambientes independientes, o de los pacientes – también responde a una cuestión evolutiva importante: la evolución es repetible? ¿La cepa evoluciona de la misma manera en diferentes personas, o que los investigadores encuentran diez resultados completamente diferentes?
Si ciertos tipos de mutaciones son repetibles – si podemos predecir de qué manera las bacterias se va a saltar, y si hay cambios que se someten a todas las bacterias-que pueden ayudar a identificar los puntos débiles de las bacterias, que podrían ser objetivos para los nuevos tipos de las intervenciones o las drogas «, dice Steve.
«Este proyecto está tratando de resolver un gran vacío en nuestra comprensión de lo que las bacterias están haciendo dentro de los pacientes», dice Craig, «porque los métodos que se utilizan en el momento en realidad no deshacerse de ellos. Con suerte, va a ser capaz de pasar esto a los médicos más adelante en el proyecto, y empezar a mejorar los resultados para los pacientes que viven con fibrosis quística. «
evolución de la fibrosis quística, evolucion fibrosis quistica, Fibrosis quística evolución, evolucion de fibrosis quística, etapas de la fibrosis quistica, tiempo de evolucion fibrosis quistica